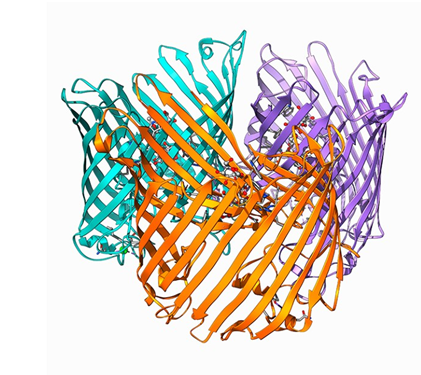
Imágen: Una forma de proteína llamada barril-β. Crédito: Laguna Design/Biblioteca de fotografías científicas
'Una caja de Pandora': el mapa de familias de estructuras de proteínas deleita a los científicos
Formas nunca vistas y conexiones inesperadas entre proteínas reveladas mediante el análisis de sus formas.
Ewen Callaway
El universo proteico acaba de volverse mucho más brillante.
Los investigadores han extraído una base de datos que contiene las estructuras de casi todas las proteínas conocidas: más de 200 millones de entradas predichas utilizando la revolucionaria red neuronal AlphaFold de Google DeepMind . El trabajo ha descubierto formas completamente nuevas, conexiones sorprendentes en la maquinaria de la vida y otros conocimientos que habrían sido impensables hace unos años.
“Gracias a AlphaFold ahora podemos explorar familias enteras de proteínas de las que no sabíamos nada”, dice Eduard Porta Pardo, biólogo computacional del Instituto de Investigación contra la Leucemia Josep Carreras (IJC) en Barcelona, España, que no participó en los estudios publicados el 13 de septiembre en Nature .
El año pasado, Google DeepMind utilizó AlphaFold para predecir la estructura de casi todas las proteínas conocidas de organismos con datos genómicos, acumulando unos 214 millones de estructuras en la base de datos AlphaFold, alojada en el Instituto Europeo de Bioinformática (EMBL-EBI) del Laboratorio Europeo de Biología Molecular en Hinxton, Reino Unido.
Grupos de estructuras
Los científicos encontraron el recurso útil al instante, pero muchos de ellos observaron sólo una única estructura, o familia de estructuras relacionadas, dice Martin Steinegger, biólogo computacional de la Universidad Nacional de Seúl, que estaba interesado en mapear las relaciones de toda la base de datos. “Pensé que sería interesante ver cuán grande es realmente nuestro universo estructural”.
Para hacer esto, un equipo codirigido por Steinegger y el biólogo computacional Pedro Beltrao, en ETH Zúrich, Suiza, desarrolló una herramienta que rápidamente podía comparar cada estructura en la base de datos, basándose en similitudes en su forma. Esto identificó más de 2 millones de "grupos" de proteínas de formas similares en la base de datos AlphaFold (1).
Los investigadores han hecho tradicionalmente este tipo de comparaciones utilizando secuencias de proteínas, que están codificadas por genes. Pero las secuencias de proteínas tienden a cambiar más rápidamente a lo largo del tiempo evolutivo, en comparación con sus estructuras, lo que limita la capacidad de encontrar proteínas relacionadas muy lejanamente. Steinegger estima que al comparar las estructuras de las proteínas, han identificado 10 veces más grupos de proteínas relacionadas que si usaran solo secuencias.
Los investigadores apenas han comenzado a explorar estas "galaxias" recientemente identificadas en el universo proteico, pero ya han descubierto algunas conexiones sorprendentes. Por ejemplo, descubrieron que una proteína que los humanos y otros organismos complejos utilizan para detectar ADN viral y desencadenar un ataque inmunológico rápido, también está en un grupo con proteínas de bacterias unicelulares y arqueas, un vínculo que no se conocía antes, dice Steinegger.
No se sabe casi nada sobre más de un tercio de los grupos de proteínas. “Realmente espero que los biólogos arrojen algo de luz sobre esta oscuridad”, dice Steinegger.
Forma nunca vista
Un segundo equipo adoptó un enfoque ligeramente diferente para iluminar la materia oscura del universo proteico. Los biólogos computacionales Joana Pereira, Janani Durairaj y Torsten Schwede, de la Universidad de Basilea, en Suiza y el Instituto Suizo de Bioinformática SIB, y sus colegas, crearon una red que conectó más de 50 millones de las estructuras predichas con mayor precisión en la base de datos AlphaFold (la herramienta proporciona una medida de qué tan buenas cree que son sus predicciones). Luego utilizaron estas agrupaciones para identificar algunos de los rincones más oscuros del universo proteico2 .
Una sorpresa agradable fue una forma de proteína que nunca se había visto. Los investigadores la denominaron “flor Beta” porque las estructuras contienen una serie de curvas cerradas (que se encuentran en una forma de proteína conocida llamada barril Beta) que se asemejan a los pétalos de una flor. Las proteínas que contienen flores Beta están relacionadas lejanamente entre sí, pero no está claro qué hacen, dice Pereira, quien está estudiando la forma más a fondo.
“Este trabajo en realidad abrió una caja de Pandora de proyectos. Tenemos que decidir cuál priorizar”, añade Pereira. Ella y sus colegas esperan que otros investigadores utilicen su red para ver cómo encaja su proteína favorita en el universo más amplio de moléculas.
Christine Orengo, bióloga computacional del University College de Londres, está entusiasmada por tener nuevas formas de navegar por el universo proteico. Pero advierte que algunas predicciones de AlphaFold consideradas muy precisas para una proteína completa pueden representar con menos precisión las formas de las porciones funcionales de la proteína, o dominios, que interesan a los investigadores. Dejar esas anomalías de lado aún debería dejar a los investigadores con un tesoro escondido. de nuevas familias de proteínas, dice Orengo, “lo cual es increíblemente emocionante”.
John Jumper, que dirige el equipo AlphaFold en Google DeepMind y no participó en ninguno de los estudios, está entusiasmado de ver a los investigadores desarrollar nuevas formas de explorar el universo que él y sus colegas desataron. Considera que estos estudios son el comienzo de un nuevo campo en el que se estudian las estructuras de las proteínas a una escala impensable. "Espero que pronto veamos más".
doi: https://doi.org/10.1038/d41586-023-02892-z
Referencias
1. Barrio-Hernández, I. et al. Nature. https://doi.org/10.1038/s41586-023-06510-w (2023)
2. Durairaj, J. et al. Nature. https://doi.org/10.1038/s41586-023-06622-3 (2023).
