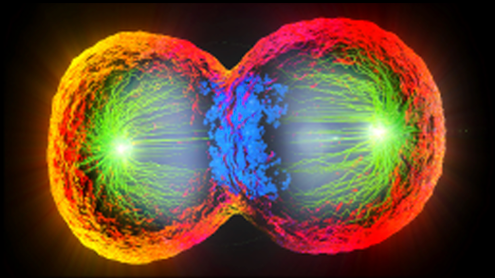
Un Atlas Celular que brinda información sobre las Proteínas Mitóticas
10 de Septiembre de 2018 | por Michele Wilson, PhD and Ruairi J Mackenzie. Escritores Científicos para Technology Networks.
Un nuevo modelo computacional 4D que integra la tecnología CRISPR, la microscopía confocal y el aprendizaje maquinal permite a los investigadores estudiar la dinámica de las proteínas específicas que impulsan la mitosis. El modelo es un marco de trabajo que puede adaptarse para estudiar otras funciones celulares.
La mitosis es una función biológica esencial que se basa en la intrincada coordinación del ensamblaje proteico en el momento adecuado. Mientras que la imagen in vivo de la célula puede revelar la distribución y la dinámica de las proteínas, el nuevo marco computacional, apodado el Atlas de la célula mitótica, integra la información y permite la cuantificación de interacciones dinámicas entre los aspectos moleculares de la maquinaria que impulsa la mitosis.
Un nuevo estudio ilustra cómo la exploración y la minería de datos de este Atlas puede ser utilizada para desarrollar nuevas hipótesis mecanísticas sobre la función de las proteínas en el interior de la célula. Los hallazgos se publicaron recientemente en la revista Nature y la investigación se llevó a cabo como un esfuerzo de colaboración entre el Laboratorio Europeo de Biología Molecular (EMBL) de Alemania y el Instituto de Investigación de Patología Molecular de Viena.
|
¿Qué es la mitosis? La progresión vital de una célula sigue naturalmente las etapas de crecimiento, supervivencia y replicación. Esa fase final es donde entra la mitosis. La división de una célula en dos no es una tarea fácil y el proceso se divide en múltiples etapas: interfase, profase, prometafase, metafase, anafase y telofase. Durante estas etapas, una red de proteínas debe organizar y separar cuidadosamente el material genético de la célula, que se divide uniformemente en dos células "hijas". Es esencial que el material genético de las células progenitoras e hijas sean idénticos, por lo que las proteínas involucradas tienen que ser bastante precisas. |
La Dra. Stephanie Alexander, Directora de investigación de EMBL en Heidelberg y una de los autores, explicó el significado de este estudio: "Nuestro estudio ofrece a la comunidad de científica, las herramientas experimentales y computacionales que permiten la gestación de un Atlas de dinámico de las proteínas implicadas.” Esto se demostró con el ejemplo de la mitosis y varias proteínas que se mostraron relevantes para la mitosis en anteriores estudios de silenciamiento génico.
Para desarrollar el programa, los investigadores generaron un modelo 4D de los cambios morfológicos que ocurren durante la mitosis en las células humanas. Imágenes fluorescentes de proteínas mitóticas knocked-in1 de células HeLa fueron capturadas y los datos fueron acondicionados usando un “tiempo mitótico estándar” basado en los cambios en la estructura del cromosoma. El establecimiento de este "tiempo estándar" permitió a los investigadores mapear objetivamente todas las imágenes celulares, en relación a una referencia de tiempo constante para el promedio.
Se seleccionaron 28 proteínas y se realizó un seguimiento mediante microscopía confocal 3D, para ver dónde se ubicaban en cada punto de tiempo dentro de la célula. Alexander explica el fundamento seguido para la selección de estas proteínas para el conjunto de datos piloto: "Básicamente empezamos con aquellas proteínas que para nosotros aparecían como más relevantes para la mitosis y que habían sido bien caracterizadas previamente. Esto se hizo principalmente porque nuestro enfoque necesitaba recuperar los conocimientos existentes y ser validado (lo que hizo). Sin embargo, sorprendentemente, nuestro enfoque también recupera nuevos aspectos o detalles que no se habían descrito antes.”
|
» https://youtu.be/zM66CvNdlds Enlace del video de cinco proteínas diferentes que son seguidas durante la división celular (de metafase a telofase): AURKB (roja), NUP107 (verde), CENPA (púrpura), CEP192 (amarilla), y TUBB4B (cian). El video representa lo que los usuarios podrían crear por sí mismos utilizando la homepage del Atlas de células mitóticas. Crédito: Arina Rybina y Julius Hossain, Ellenberg group, EMBL. |
El equipo desarrolló un enfoque de aprendizaje maquinalsupervisado para definir estructuras subcelulares. También entrenaron un modelo de regresión para asignar cantidades de proteína a diferentes compartimentos de referencia, lo que les permitió comparar cuantitativamente los flujos en los niveles específicos de proteína.
En clonación molecular, un Knock-in se refiere a un método de ingeniería genética que involucra la introducción de un ADNc en un locus particular de un cromosoma del organismo.
Los autores prevén que su marco de trabajo computacional puede adaptarse a otras funciones biológicas esenciales, como la migración celular o la diferenciación celular. "el concepto de estandarizar el contexto celular espacio-temporal para analizar las distribuciones dinámicas de proteínas para entender los procesos celulares presentados aquí, es genérico y visualizamos que puede ser adaptado a otras funciones biológicas esenciales", escribieron los autores.
El modelo proporciona un enfoque nuevo y objetivo para entender una gama de procesos celulares, como se describe en la discusión: "Nuestro modelo provee un sistema de referencia espacio-temporal estandarizado pero dinámico para la célula mitótica, que puede ser usado para integrar información cuantitativa sobre cualquier cantidad de distribuciones proteicas muestreadas en miles de experimentos de células individuales diferentes."
Alexander elabora: "Estas herramientas nos permiten explorar posibles interacciones proteicas y redes funcionales. Debido a que este Atlas es dinámico, podemos entender cómo la información se transmite durante la mitosis y cómo cambian las interacciones proteicas. Esto permite formular hipótesis dirigidas para historias de seguimiento detalladas... Verlo todo junto al final, similar a un gran rompecabezas, fue muy emocionante.”
Para permitir futuros estudios, los métodos experimentales, la plataforma de microscopía cuantitativa y el código para crear Atlas de proteínas dinámicas ahora están abiertamente disponibles para que otros los utilicen. Alexander explica cómo este Atlas se puede emplear para entender los patrones celulares: "Una vez que el atlas de proteínas se complete, es decir, se integren los datos de más proteínas, podremos estudiar las redes de proteínas e identificar los centros donde se reúne toda la información, o también donde existen vías alternativas. Nuevamente, como nuestro atlas cubre el progreso mitótico, también podremos ver si un eje es central para todo el proceso o si los ejes cambian con el tiempo. Entonces, podemos usar el atlas de células mitóticas para identificar los puntos vulnerables de todo el sistema.”
» Más información: http://www.mitocheck.org/mitotic_cell_atlas/
Referencia:
Cai, Y., Hossain, M. J., Hériché, J., Politi, A. Z., Walther, N., Koch, B.,... Ellenberg, J. (2018). Experimental and computational framework for a dynamic protein atlas of human cell division. Nature: doi:10.1038/s41586-018-0518-z
Abstract (original)
Essential biological functions, such as mitosis, require tight coordination of hundreds of proteins in space and time. Localization, the timing of interactions and changes in cellular structure are all crucial to ensure the correct assembly, function and regulation of protein complexes1,2,3,4. Imaging of live cells can reveal protein distributions and dynamics but experimental and theoretical challenges have prevented the collection of quantitative data, which are necessary for the formulation of a model of mitosis that comprehensively integrates information and enables the analysis of the dynamic interactions between the molecular parts of the mitotic machinery within changing cellular boundaries. Here we generate a canonical model of the morphological changes during the mitotic progression of human cells on the basis of four-dimensional image data. We use this model to integrate dynamic three-dimensional concentration data of many fluorescently knocked-in mitotic proteins, imaged by fluorescence correlation spectroscopy-calibrated microscopy5. The approach taken here to generate a dynamic protein atlas of human cell division is generic; it can be applied to systematically map and mine dynamic protein localization networks that drive cell division in different cell types, and can be conceptually transferred to other cellular functions.
Resumen (Original)
Las funciones biológicas esenciales, como la mitosis, requieren una estrecha coordinación en el espacio y el tiempo de cientos de proteínas. La localización, el momento de sus interacciones y los cambios en la estructura celular son cruciales para asegurar el correcto ensamble, así como la función y regulación de los complejos proteicos1,2,3,4. La imagen de las células vivas puede revelar distribuciones y dinámicas proteicas, pero los desafíos experimentales y teóricos han impedido la recopilación de datos cuantitativos, necesarios para la formulación de un modelo de mitosis que componga integralmente la información y permita el análisis de las interacciones dinámicas entre las partes moleculares de la maquinaria mitótica en los límites celulares cambiantes. Aquí generamos un modelo canónico de los cambios morfológicos que suceden durante la progresión mitótica de células humanas basados en datos de imágenes de cuatro dimensiones. Utilizamos este modelo para integrar datos tridimensionales dinámicos de la concentración de varios proteínas mitóticas fluorescentemente knocked-in, imágenes obtenidas por microscopia de fluorescencia calibrada con espectroscopia de correlación5. El enfoque que se toma aquí para generar un Atlas de proteínas dinámicas de la división celular humana es genérico; puede aplicarse sistemáticamente para mapear y realizar minería de las redes de localización de proteínas dinámicas que conducen la división celular en diferentes tipos celulares, y asimismo pueden ser transferidas conceptualmente a otras funciones celulares.
