Uso de aprendizaje automático para crear mejores terapias génicas
12 de febrero de 2021 |Historia de Original del Instituto Wyss
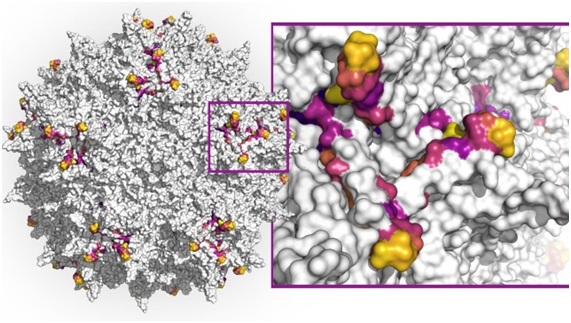
En su estrategia basada en aprendizaje automático de diversificación de la cápside, el equipo se centró en un péptido de 28 aminoácidos de un segmento de la proteína de la cápside AAV2 VP3 que expone la cápside AAV a anticuerpos neutralizantes producidos por los individuos y por lo tanto puede ser la causa de una respuesta inmune contra el virus. Las porciones de color púrpura de este péptido están enterradas más profundamente en la cápside, mientras que las partes amarillas se exponen en la superficie del virus. Crédito: Wyss Institute de la Universidad de Harvard (original por Drew Bryant).
Los adenovirus asociados (AAVs) se han convertido en vehículos prometedores para la entrega a tejidos defectuosos de terapias génicas en el cuerpo humano, porque no son patógenos y pueden transferir ADN terapéutico a las células diana. Sin embargo, mientras que los primeros productos de terapia génica aprobados por la Administración Federal de Medicamentos (FDA) utilizan vectores AAV, todavía no han alcanzado todo su potencial para hacer frente a los desafíos terapéuticos genéticos.
En primer lugar, las cápsides de AAV utilizadas actualmente -las estructuras proteicas esféricas que envuelven el genoma de ADN de una sola hebra del virus, que se puede modificar para codificar genes terapéuticos- están limitados en su capacidad de mejorar específicamente el tejido afectado por una enfermedad y su distribución más amplia por todo el cuerpo humano, hace que se diluyan. En segundo lugar, los sistemas inmunitarios de los pacientes, después de haber estado expuestos a un virus similar del AAV, pueden producir anticuerpos neutralizantes que, incluso en niveles bajos, pueden destruir los AAVs tras la reexposición (neutralización), bloqueando la entrega de sus cargas terapéuticas de ADN.
Para superar este problema de neutralización, los investigadores están diseñando cápsides de AAV mejorados que se espera sean capaces de evadir el sistema inmune. Los métodos utilizados actualmente, incluidas las estrategias de “evolución dirigida” que aceleran la evolución de una proteína en condiciones de laboratorio, sólo pueden crear una diversidad limitada de cápsides, la mayoría de ellas, todavía parecidos a las variantes de AAV naturales conocidas como serotipos. Sin embargo, sigue siendo difícil generar suficiente diversidad utilizando este enfoque sin perder otras funciones deseadas de la cápside, como su estabilidad o capacidad para unirse a tipos celulares específicos.
Ahora, un nuevo estudio iniciado por el equipo de Biología Sintética liderado por George Church,miembro del Instituto Wyss para la Ingeniería Biológicamente Inspirada de Harvard, e impulsado por una colaboración con Google Research ha aplicado un enfoque computacional de aprendizaje profundo para diseñar variantes de cápsides muy diversas del serotipo AAV2 a través de codificaciones de ADN de un segmento clave de estas proteínas que juega un papel en el reconocimiento inmune, así como la infección de los tejidos objetivo. El AAV2 es el serotipo más estudiado y se ha utilizado en la primera terapia génica aprobada por la FDA, para tratar una enfermedad cegadora.
A partir de una colección relativamente pequeña de datos de cápsides, el equipo entrenó múltiples métodos de aprendizaje automático y los utilizó para diseñar 200.000 variantes de virus. 110.689 de estas variantes produjeron virus AAV viables. Entre dos serotipos AAV naturales, en los que se esperaba que 12 aminoácidos dentro de este segmento difirieran. El esfuerzo del equipo produjo más de 57.000 variantes que mostraron una diversidad mucho mayor que esta, algunas que contenían hasta 29 aminoácidos sustituidos combinados o insertados adicionalmente. Los hallazgos se publicaron en Nature Biotechnology.
“Nuestro enfoque logra mayor diversidad funcional que cualquier biblioteca de cápsides conocida. Desbloquea vastas áreas de espacio de secuencia funcional que antes eran inalcanzables, con muchas aplicaciones potenciales para generar vectores virales mejorados, como los AAVs con una inmunogenicidad mucho más reducida y una selectividad del tejido objetivo mucho mejor, y también para terapias génicas altamente eficientes”, dijo el autor de correspondencia Eric Kelsic, quien inició el proyecto con Church, y cofundó la startup DynoTherapeutics, donde ahora es CEO. La misión de DynoTherapeutics es desarrollar vehículos avanzados de administración de terapia génica mediante el empleo de enfoques de vanguardia de inteligencia artificial (IA).
Utilizando múltiples estrategias de diseño, el equipo primero generó conjuntos de datos más pequeños en los que podían entrenar varios modelos de aprendizaje automático. Se trataba de colecciones de cápsides de AAV con un número variable de mutaciones introducidas en un segmento de 28 aminoácidos de la proteína AAV2 VP3 que forma parte de la cápside y la expone a anticuerpos neutralizantes. Un método de alto rendimiento que permite la síntesis de secuencias de cápsides mutadas y experimentos in vitro para pruebas que produjeron cápsides estables viables, proporcionó un banco de pruebas altamente eficaz para su enfoque general. Los resultados de este primer estudio experimental fueron utilizados por el equipo como datos de entrenamiento para tres modelos alternativos de aprendizaje automático que generaron un número mucho mayor de diversas variantes cápsides para ser probados con un experimento de validación final.
Un cuello de botella central en la creación de diversas cápsides y variantes de AAV que pueden evadir la neutralización, es la producción de cápsides que permanecen estables: la mayoría de las variantes no se ensamblarán en cápsides funcionales o empaquetarán sus genomas de AAV. “Los modelos de red neuronal profunda que implementamos con nuestros colaboradores de Google predijeron con precisión la viabilidad de los cápsides en variantes extremadamente diversas. Alcanzar este nivel de diversidad en el segmento de las cápsides es un hito importante sobre el que podemos aprovecharnos para encontrar cápsides para la terapia génica que evaden el sistema inmunitario”, dijo el coautor Sam Sinai, un exestudiante graduado de Church que se unió al equipo de Kelsic en el Instituto Wyss y es cofundador al frente del equipo de aprendizaje automático de DynoTherapeutics. “Ahora podemos tomar enfoques similares para crear cápsides AAV con mucha mejor selectividad tisular.”
En 2019, otro equipo de Wyss que incluía aKelsic, Sinaí, y su mentor Church, publicaron en Scienceun enfoque relacionado en el que mutaron,de diferentes maneras, uno por uno cada uno de los 735 aminoácidos dentro de toda la cápside de AAV2. Lo que llamaron una búsqueda "amplia" dio lugar a una gran biblioteca de AAV que identificó cambios que afectan la viabilidad de AAV2 y su potencial de búsqueda de blancos dirigidos contra órganos específicos en ratones, así como una proteína accesoria previamente desconocida que se une a las membranas celulares y que estaba oculta dentro de la secuencia de ADN de codificación de las cápsides. En su estudio anterior, los investigadores utilizaron un modelo experimental simple para optimizar la capacidad del virusde reconocer tejidos específicos.
“Este nuevo estudio que involucra modelos de aprendizaje automático desarrollados con Google Research complementa muy bien nuestro trabajo anterior en el que se centra en una pequeña, pero muy importante, región de la cápside de AAV y con una resolución sin precedentes”, dijo el coautor para correspondencia, Church. “Muestra que las redes neuronales combinadas con las pruebas sintéticas de alto rendimiento desarrolladas en nuestro laboratorio están cambiando la forma en que diseñamos vehículos de administración de genes y fármacos proteicos”. Church es el líder de la plataforma de Biología Sintética del Instituto Wyss donde se inició el proyecto, y profesor de genética en la Escuela de Medicina de Harvard y de Ciencias de la Salud y Tecnología en Harvard y el MIT.
“Este trabajo da una idea de cómo los enfoques de inteligencia artificial, como el aprendizaje automático, están abriendo vastos nuevos espacios de diseño que permiten el desarrollo de fármacos completamente nuevos y enfoques de administración de fármacos para combatir innumerables desafíos para la salud humana. También destaca el compromiso del Instituto Wyss con la resolución de problemas computacionales en áreas donde se necesitan desesperadamente nuevas terapias”, dijo el Director Fundador de Wyss, Donald Ingber, quien también es profesor de biología vascular en la Escuela de Medicina de Harvard,del Hospital Infantil de Boston, y profesor de Bioingeniería en SEAS
Referencia:
Bryant DH, Bashir A, Sinai S, et al. Deep diversification of an AAV capsid protein by machine learning. Nat Biotechnol. 2021. doi:10.1038/s41587-020-00793-4.
Este artículo ha sido republicado a partir de los siguientes materiales. Nota: el material puede haber sido editado por su longitud y contenido. Para obtener más información, ver la fuente citada.
