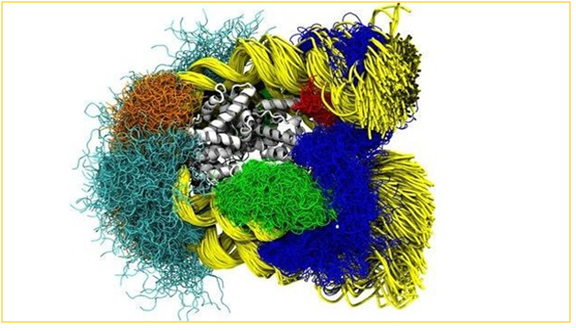
Tres microsegundos del tiempo de vida de un nucleosoma: Las instantáneas temporales se tomaron cada 4 nanosegundos y se superponen a la región central de las histonas (blancas). Se muestran las posiciones del ADN y las colas flexibles de las histonas en todas las instantáneas. El ADN está en amarillo y las colas de las histonas en azul, verde, rojo, naranja y cian. El amplio movimiento de los brazos de ADN se conoce como movimiento de respiración de los nucleosomas. Sorprendentemente, en este nucleosoma, la parte inferior del brazo se mueve más que la superior debido a la secuencia de ADN. Crédito: Jan Huertas y Vlad Cojocaru; MPI Münster / Instituto Hubrecht.
Los Movimientos de los Nucleosomas Capturados en Detalle Atómico
10 de junio de 2021 | Historia original del Instituto Max Planck de Biomedicina Molecular en Münster
Investigadores del Instituto Hubrecht de Utrecht (Países Bajos) y del Instituto Max Planck de Biomedicina Molecular de Münster (Alemania) utilizaron simulaciones por computadora para revelar con detalle atómico cómo se abre una corta pieza de ADN mientras está estrechamente envuelta alrededor de las proteínas que empaquetan nuestro genoma. Estas simulaciones proporcionan información sin precedentes sobre los mecanismos que regulan la expresión génica. Los resultados fueron publicados en PLoS Computational Biology.
Cada célula de nuestro cuerpo contiene dos metros de ADN. Con el fin de encajar todo el ADN en el pequeño núcleo de la célula, el ADN está estrechamente empaquetado en una estructura conocida como cromatina. La cromatina es una matriz de estructuras idénticas más pequeñas llamadas nucleosomas. En cada nucleosoma, el ADN se envuelve alrededor de 8 proteínas llamadas histonas. La cromatina no es uniformemente compacta en todo el genoma. La estanqueidad del envase es importante para regular qué genes se expresan y, por lo tanto, qué proteínas son producidas por una célula.
Las transiciones de ADN herméticamente a holgadamente empaquetado –de cromatina cerrada a abierta– son esenciales para que las células se conviertan a otro tipo de célula. Estas conversiones celulares son sellos distintivos del desarrollo y la enfermedad, pero también se utilizan a menudo en terapias regenerativas. La comprensión de cómo ocurren tales transiciones puede contribuir a la comprensión de enfermedades y a optimizar conversiones terapéuticas del tipo de la célula.
Nanoscopio computacional
Un paso en la apertura de la cromatina es el movimiento del ADN mientras está envuelto en nucleosomas. Como todas las estructuras moleculares de nuestras células, los nucleosomas son dinámicos. Se mueven, se retuercen, “respiran”, se desenvuelven y se envuelven nuevamente. Visualizar estos movimientos usando métodos experimentales es a menudo muy difícil. Una alternativa es utilizar el llamado "nanoscopio computacional".
Los investigadores utilizan el término nanoscopio computacional para referirse a un conjunto de métodos de simulación por computadora. Estos métodos les permiten visualizar los movimientos de las moléculas a lo largo del tiempo. En los últimos años, los métodos se han vuelto tan precisos que los investigadores comenzaron a referirse a ellos como un nanoscopio computacional; observar las moléculas que se mueven en la computadora es similar a observarlas bajo un nanoscopio de muy alta resolución.
“Respiración” de nucleosomas
Jan Huertas y Vlad Cojocaru, apoyados por Hans Schöler del Instituto Max Planck de Biomedicina Molecular (Münster, Alemania), generaron múltiples películas en tiempo real de los movimientos de los nucleosomas, cada uno cubriendo un microsegundo de la vida útil del nucleosoma. Usando estas películas, monitorearon cómo los nucleosomas se abren y cierran en un movimiento conocido como “respiración” de los nucleosomas.
En su nuevo artículo, publicado en PLoS Computational Biology, Huertas y Cojocaru describen qué causa la “respiración” de los nucleosomas. En primer lugar, descubrieron que el orden en que se disponen los bloques de construcción del ADN –la secuencia de ADN– es importante para la respiración de los nucleosomas. En segundo lugar, la dinámica de las colas de histonas es esencial para este proceso. Estas colas de histonas son regiones flexibles en las histonas que desempeñan un papel en la regulación de la expresión génica. Si bien el papel de las colas de histonas se ha estudiado intensamente, poco se sabe sobre cómo influyen en los movimientos de los nucleosomas individuales. Con sus simulaciones, Huertas y Cojocaru describieron la relación entre las colas de histonas y la respiración de nucleosomas en detalle atómico.
Modificaciones de las histonas
“Poder observar la respiración de nucleosomas en simulaciones por computadora es muy desafiante. El hecho de que ahora hayamos podido visualizar esto representa un paso importante hacia la simulación del espectro completo de la dinámica del nucleosoma, desde la respiración hasta el desenvolvimiento. También nos permite estudiar cómo estos movimientos se ven afectados por las modificaciones de las histonas, que se producen en diferentes células y regiones de nuestro ADN. Nuestras simulaciones revelaron que dos colas de histonas son responsables de mantener el nucleosoma cerrado. Solo cuando estas colas flexibles se alejaron de regiones particulares del ADN, el nucleosoma pudo abrirse", afirmó el líder de la investigación, Vlad Cojocaru.
Jan Huertas, primer autor de la publicación y recién doctorando, añade: “La cromatina activa (abierta) e inactiva (cerrada) contiene diferentes modificaciones de las colas de histonas. El siguiente paso es realizar simulaciones con tales modificaciones. La resolución atómica de las simulaciones nos permitiría identificar cómo afecta cada modificación a los nucleosomas y a la dinámica de la cromatina”.
Hacia la comprensión de la epigenética
Los tres investigadores están entusiasmados con el futuro del uso de simulaciones atomísticas por computadora en la comprensión de los mecanismos de expresión génica en el desarrollo y la enfermedad. “Con el aumento adicional de la potencia computacional disponible en el mundo, pronto podremos simular milisegundos de una vida útil de nucleosoma con todos sus átomos incluidos. Además, podremos simular rutinariamente múltiples nucleosomas para estudiar el efecto de diferentes modificaciones de las histonas sobre la expresión génica. Esto dará una visión sin precedentes de los mecanismos que regulan la expresión génica”, concluye Cojocaru.
Referencia
Huertas J, Schöler HR, Cojocaru V. Histone tails cooperate to control the breathing of genomic nucleosomes. PLOS Computational Biology. 2021; 17 (6): e1009013.
doi: 10.1371/journal.pcbi.1009013.
Este artículo ha sido republicado a partir de los siguientes materiales. Nota: el material puede haber sido editado por su longitud y contenido. Para más información, ver la fuente citada.
