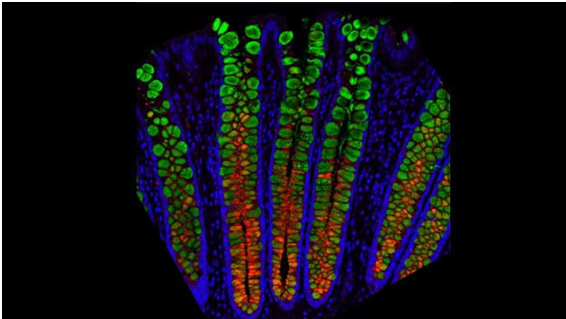
Imagen: Proteína MUC5b (roja) en células caliciformes residentes en cripta; MUC2 (verde) en todas las células caliciformes. Crédito: Magness Lab, Facultad de Medicina de la UNC
Mapeo del intestino humano con resolución celular
21 de febrero de 2022| Historia original de University of North Carolina Health
Si te pones nervioso, es posible que lo sientas en tu intestino. Si se ingiere ají picante, tu intestino puede rebelarse, pero tu amigo puede comer cualquier cosa y sentirse bien. Puede tomar ibuprofeno como un caramelo sin efectos nocivos, pero el estómago de otro amigo podría sangrar y podría no aliviar el dolor. ¿Por qué? La respuesta rápida es porque todos somos diferentes. Las siguientes preguntas son ¿quétan diferentes exactamente, y qué significan estas diferencias para la salud y la enfermedad? Responder a esto es mucho más difícil, pero el laboratorio de Scott Magness, PhD de la Facultad de Medicina de la UNC, está revelando algunas respuestas científicas interesantes.
Por primera vez, el laboratorio de Magness utilizó tractos gastrointestinales humanos completos de tres donantes de órganos para mostrar cómo los tipos de células difieren en todas las regiones de los intestinos, para arrojar luz sobre las funciones celulares y para mostrar las diferencias de expresión génica entre estas células y entre los individuos.
Este trabajo, publicado en Cellular and Molecular Gastroenterology and Hepatology, abre la puerta a explorar las muchas facetas de la salud intestinal de una manera mucho más precisa y con mayor resolución que nunca.
“Nuestro laboratorio demostró que es posible aprender sobre la función de cada tipo de célula en procesos importantes, como la absorción de nutrientes, la protección contra parásitos y la producción de mucus y hormonas que regulan el comportamiento alimentario y la motilidad intestinal”, explicó Magness, profesor asociado en el Departamento de Ingeniería Biomédica del Estado Conjunto UNC-NC y autor principal del artículo. “También aprendimos cómo el revestimiento intestinal podría interactuar con el medio ambiente a través de receptores y sensores, y cómo los medicamentos podrían interactuar con diferentes tipos de células”.
El intestino sensible
Piense en una voz en off de un comercial farmacéutico típico, cuando el relator recita agradablemente posibles efectos secundarios, como diarrea, vómitos, sangrado intestinal y otros daños colaterales desagradables. Bueno, el laboratorio Magness está tratando de entender por qué ocurren esos efectos secundarios, hasta el nivel de las células individuales, sus funciones, sus ubicaciones y sus genes.
Para esta investigación, el laboratorio Magness se centró en el epitelio: una capa gruesa de una sola célula que separa el interior de los intestinos y el colon de todo lo demás. Al igual que otras poblaciones celulares y la microbiota, el epitelio es increíblemente importante para la salud humana, y durante años los científicos lo han estado explorando. Pero hasta ahora, los investigadores solo podían tomar pequeñas biopsias del tamaño de granos de arroz de algunas partes del tracto digestivo, generalmente del colon o regiones limitadas del intestino delgado.
“Tal exploración sería como mirar a Estados Unidos desde el espacio, pero solo investigar lo que está sucediendo en Massachusetts, Oklahoma y California”, dijo Magness. “Para aprender realmente sobre el país, nos gustaría ver todo”.
Magness se apoyó en los coautores, el becario postdoctoral Joseph Burclaff, PhD, y el estudiante graduado, Jarrett Bliton, ambos aprendices en el laboratorio de Magness.
“No solo queremos identificar dónde se encuentran las células, sino que queremos saber exactamente qué tipos de células hacen qué y por qué”, analizó Burclaff. “Entonces, siguiendo con la analogía del mapa, no queremos simplemente decir: 'oh, está Carolina del Norte'. Queremos saber dónde conseguir el mejor asado. Queremos una vista a nivel del suelo para saber tanto como sea posible".
En el pasado, los investigadores mezclaban esas biopsias del tamaño de un arroz para identificar todos los tipos de células epiteliales y aprender algunas características generales de estas células. El enfoque de Magness fue tomar muestras de miles de células individuales de cada parte del tracto digestivo inferior (intestino delgado y colon) para crear un atlas y luego estudiar las funciones potenciales de estas células a través de los genes que expresa cada célula. Saber todo esto profundizaría el conocimiento científico sobre el epitelio intestinal y, con suerte, alentaría a otros científicos a explorar la función biológica de cada célula, tanto en las enfermedades como en el desafortunado escenario de efectos secundarios provocado por los fármacos.
Para hacer una inmersión celular individual tan profunda, Magness necesitaba dos cosas: una mejor tecnología y todo el tracto digestivo de los humanos.
La biología de los datos
UNC-Chapel Hill adquirió tecnología de secuenciación de ARN de última generación hace varios años para la creación de la Instalación Central de Análisis Avanzado a través del Centro de Enfermedades Gastrointestinales y Biología de la UNC, que desarrolló el peso científico e intelectual (profesores de investigación, personal, postdoctorados y estudiantes) para usar equipos de última generación.
El grupo de Magness adquirió tractos digestivos humanos a través de un acuerdo de investigación con los servicios de donantes de órganos en HonorBridge.
Cuando los intestinos se colectan para trasplante y, si no son reclamados por grupos de mayor prioridad, el personal de HonorBridge coordina con el grupo de Magness para donar los órganos de grado de trasplante para la investigación.
De seis a ocho horas después de la colecta, el laboratorio de Magness recibe tractos intestinales intactos, cada uno de aproximadamente 4,572 a 9,144metros de largo. Eliminan la capa epitelial, que es una pieza larga y conectada de tejido a pesar de tener sólo una célula de grosor. Luego, los investigadores usan enzimas para descomponer el epitelio en células individuales. Para este estudio, repitieron esto para órganos de tres donantes separados.
Utilizando la tecnología de secuenciación para caracterizar la expresión génica, el grupo de Magness primero extrajoARN de cada célula, manteniéndolas separadas, y luego las secuenciaron, reflejando una toma instantánea de qué genes está expresando cada célula intestinal y cuánto.
“La imagen que obtenemos de cada célula es un mosaico de todos los diferentes tipos de genes que producen las células y este complemento de genes crea una 'firma' para decirnos qué tipo de célula es y potencialmente qué está haciendo”, explica Magness. ¿Es una célula madre o una célula mucosa o una célula productora de hormonas o una célula inmuno-señalizadora?
Burclaff agregó: “Pudimos ver las diferencias en los tipos de células en todo el tracto digestivo, y podemos ver diferentes niveles de expresión génica en los mismos tipos de células de tres personas diferentes. Podemos ver los diferentes conjuntos de genes activados o desactivados en células individuales. Así es como, por ejemplo, podríamos comenzar a entender por qué algunas personas se intoxicancon ciertos alimentos o medicamentos y otras personas no".
Un problema importante con este tipo de investigación es la gran cantidad de datos producidos. La secuenciación de cada célula recoge alrededor de 11.000 "lecturas", o muestras individuales de productos genéticos en una sola célula, y en muchos miles de células individuales, cada una con diferentes combinaciones de los más de 20,000 genes humanos que se activan o desactivan. Esto crea casi 140.000.000 de datos puntuales para la totalidad de las 12.590 células en el estudio que deben colocarse en un formato visualizable para que los científicos puedan dar sentido a la gran cantidad de información.
“El cerebro humano sólo permite comprender dos dimensiones, tres es un desafío”, explica Magness. “Si se le agregala dimensión temporal, es aún más difícil comprender lo que una sola célula está haciendo. La cantidad de datos que produjeron nuestros experimentos fue básicamente millones de dimensiones a la vez”.
Bliton ideó técnicas computacionales para filtrar los datos y producir un conjunto de datos manejable que incluía poblaciones celulares de todas las partes del tracto gastrointestinal. Luego, basándose en lo que Magness y otros investigadores ya habían aprendido de cada tipo de célula, pudo identificar computacionalmente cada tipo de célula de cada región. Luego representó estos datos de una manera que los humanos pueden entender e interpretar.
Controlar el inmenso volumen de datos permitió a los científicos aprender mucho sobre cada tipo de célula. Considere las célulaspenacho, descubiertas hace 40 años. Resulta que estas células expresan genes similares a los de las papilas gustativas de la lengua. Otros investigadores descubrieron que estas células penacho, detectaban infecciones por gusanos y enviaban señales al sistema inmunológico para comenzar a librar una guerra. El laboratorio de Magness mostró que las células penacho exhiben un conjunto de genes que se cree que son importantes para detectar y "probar" otros tipos de contenido intestinal para que puedan señalizar al sistema inmunológico si es necesario. Esto representaría una función mucho más amplia que detectar si hay un parásito en su intestino o no.
“No sólo describimos cada tipo celular y cada gen que se expresa individualmente, sino que también observamos las funciones potenciales”, dijo Burclaff. “Si nos fijamos en el mucus intestinal, que es una mezcla compleja que protege las células, mostramos qué células expresan varias proteínas de mucina, cuánto y en qué regiones del tracto digestivo. Observamos dónde se expresan las enzimas específicas que digieren los alimentos. Observamos células con expresión génica antiinflamatoria y genes de sinapsis donde el intestino probablemente está conectado a los nervios para que pueda dialogar con el resto del cuerpo. Observamos las acuaporinas, proteínas involucradas en la transferencia de agua a través de la membrana intestinal".
Lo que el grupo de Magness encontró fue un nivel completamente nuevo de variación en las funciones potenciales que no se había apreciado previamente a través de la mezcla de muestras de biopsia.
Los investigadores exploraron todos los receptores epiteliales, las proteínas de la superficie celular utilizadas para comunicarse con otras células y moléculas y con el entorno del intestino. Magness y sus colegas pudieron ver qué receptores se expresaban más y en qué tipos de células, pintando una nueva imagen de cómo las células podrían interactuar con el contenido intestinal, como nutrientes, microbios, toxinas y medicamentos.
“Hasta donde sabemos, somos los primeros en hacer este tipo de análisis a lo largo del intestino humano de tres donantes completos”, dijo Bliton. “Podemos observar cada tipo de célula y predecir qué productos farmacéuticos podrían afectar individualmente a qué tipo de célula”.
Por ejemplo, una clase de medicamentos para tratar la enfermedad inflamatoria intestinal, están diseñados para alcanzar objetivos específicos, ciertas células inmunes que desencadenan la inflamación. Pero el laboratorio de Magness aprendió que algunas células epiteliales expresan los mismos genes que los de las células inmunes que están destinadas a ser el objetivo. Este hallazgo indica que podría haber efectos “ por fuera del objetivo” en las células epiteliales que no están previstos y podrían provocar efectos secundarios.
“Esto no se sabía”, dijo Burclaff. “Muchos medicamentos tienen efectos secundarios gastrointestinales negativos. Y podría deberse a que los medicamentos están afectando a células individuales a lo largo de toda la longitud del tracto gastrointestinal. Mostramos dónde se expresan más estos receptores y en qué tipos de células”.
Este tipo de conocimiento es solo un resultado del estudio inicial del laboratorio de Magness.
“Queremos que la comunidad científica, médica y farmacéutica use lo que hemos encontrado”, dijo Magness. “Adoptamos un enfoque analítico para abordar metódicamente cada tipo celular, producir hojas de cálculo fáciles de leer y accesibles para la mayoría de los científicos, y mostrar varios ejemplos de lo que podemos descubrir con este tipo de enfoque de alta resolución y precisión”.
Referencia
Burclaff J, Bliton J, Breau KA, et al. A proximal-to-distal survey of healthy adult human small intestine and colon epithelium by single-cell transcriptomics. CMGH Cell. Mol. Gastroenterol. 2022. doi: 10.1016/j.jcmgh.2022.02.007
Este artículo ha sido republicado a partir de los siguientes materiales. Nota: el material puede haber sido editado por su longitud y contenido. Para obtener más información, ver la fuente citada.
